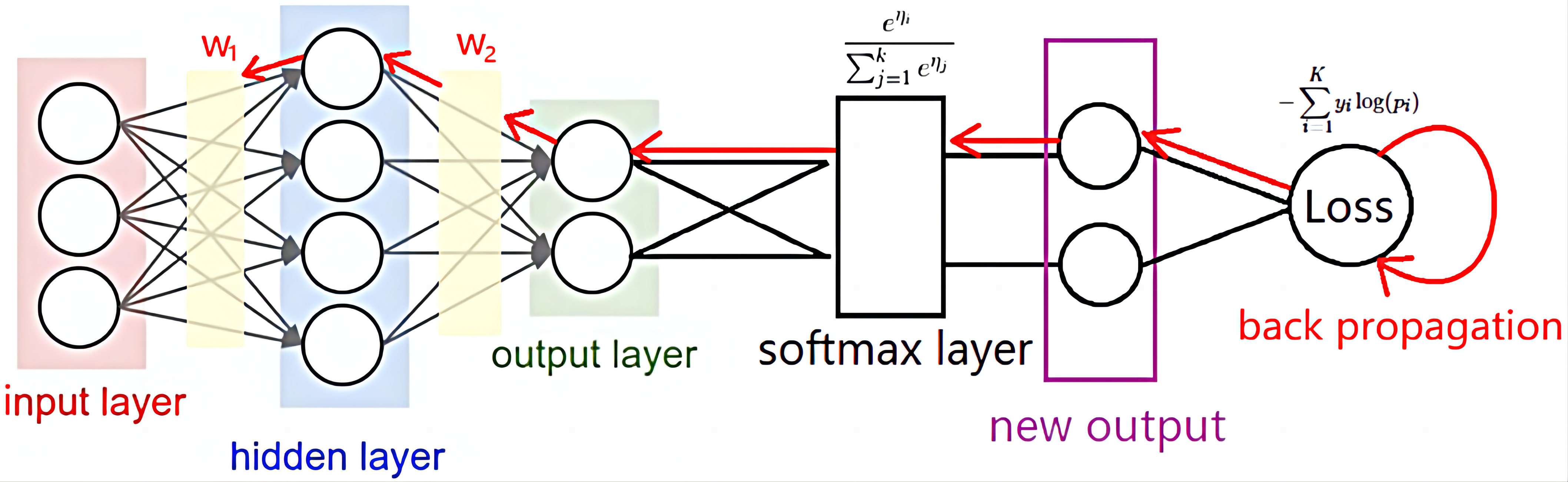
scGPT
scGPT模型及其在不同任务中的表现
作者使用的基准方法和对比方法在不同任务和指标上的表现
以下是作者在不同任务下使用的基准方法或对比方法,以及它们在相应指标上的表现总结:
单细胞RNA测序数据的细胞类型注释
- 基准方法: scBERT 和 TOSICA
- 指标: 准确性(Accuracy)、精确度(Precision)、召回率(Recall)、宏F1分数(Macro F1)
- 表现: 文章中未提供具体数值,但指出scGPT在所有分类指标上都优于这两种方法。
遗传扰动响应预测
- 对比方法: GEARS 和线性回归模型
- 指标: Pearsondelta 和 Pearsondelta on differentially expressed genes (DE genes)
- 表现: 文章中未提供具体数值,但强调scGPT在所有三个数据集上都取得了最高分,并且在预测扰动后变化的基因表达上,持续优于其他方法5-20%。
多批次scRNA-seq数据整合
- 对比方法: Seurat、Harmony 和 scVI
- 指标: AvgBIO分数(包括NMIcell、ARIcell和ASWcell)、AvgBATCH分数(包括ASWbatch和GraphConn)
- 表现: 文章中未提供具体数值,但指出scGPT在PBMC 10k数据集上的表现优于这些方法。
单细胞多组学数据整合
- 对比方法: Seurat (v.4)、scGLUE 和 scMoMat
- 指标: NMIcell、ARIcell、ASWcell、AvgBIO以及ASWbatch、GraphConn、AvgBATCH
- 表现: 文章中未提供scGLUE和scMoMat的具体数值表现,但在10x Multiome PBMC数据集上,scGPT是唯一成功生成CD8+ naive细胞独特聚类的方法。在BMMC数据集上,scGPT比Seurat (v.4)展现出更明确的聚类结构。
基因调控网络推断
- 基准方法: 基于表达相关性的共表达网络
- 指标: 未明确提及,但进行了路径富集分析以验证基因程序的质量
- 表现: 文章中未提供共表达网络的具体数值表现,但指出scGPT在所有分辨率下识别的富集路径数量显著多于共表达方法。
请注意,文章中并没有提供所有对比方法的具体数值表现,而是通过比较来强调scGPT在各个任务中的优越性能。为了获得其他方法的详细表现数据,可能需要查阅原始的基准测试或方法论文。
scGPT模型验证采用的数据集
作者在验证scGPT模型的有效性时,使用了多个数据集,并针对不同的任务和指标进行了评估。以下是使用的各个数据集、对应的任务以及评估所用的指标:
-
CELLxGENE scRNA-seq数据集:
- 任务: 用于构建全人类基础模型的预训练。
- 指标: 未明确提及,但这是构建模型的基础数据集。
-
多发性硬化症(MS)数据集:
- 任务: 细胞类型注释。
- 指标: 准确性(Accuracy)、精确度(Precision)、召回率(Recall)、宏F1分数(Macro F1)。
-
骨髓瘤(Myeloid)数据集:
- 任务: 细胞类型注释、遗传扰动响应预测。
- 指标: 同上。
-
人类胰腺(Human Pancreas)数据集:
- 任务: 细胞类型注释。
- 指标: 同上。
-
PBMC 10k数据集:
- 任务: 多批次scRNA-seq数据整合。
- 指标: AvgBIO分数(包括NMIcell、ARIcell和ASWcell)、AvgBATCH分数(包括ASWbatch和GraphConn)。
-
周围皮层(Perirhinal Cortex)数据集:
- 任务: 多批次scRNA-seq数据整合。
- 指标: 同上。
-
COVID-19数据集:
- 任务: 多批次scRNA-seq数据整合。
- 指标: 同上。
-
Adamson、Norman和Replogle数据集:
- 任务: 遗传扰动响应预测。
- 指标: Pearsondelta、Pearsondelta on differentially expressed genes (DE genes)。
-
10x Multiome PBMC数据集:
- 任务: 单细胞多组学(scMultiomic)数据整合。
- 指标: NMIcell、ARIcell、ASWcell、AvgBIO以及ASWbatch、GraphConn、AvgBATCH。
-
BMMC数据集:
- 任务: 单细胞多组学数据整合。
- 指标: 同上。
-
ASAP PBMC数据集:
- 任务: 单细胞多组学数据整合。
- 指标: 同上。
-
免疫人类(Immune Human)数据集:
- 任务: 基因调控网络推断。
- 指标: 未明确提及,但通过与已知生物学和功能组的一致性来验证网络的质量。
这些数据集覆盖了从基础模型预训练到各种下游应用的广泛任务,作者通过这些任务和指标全面评估了scGPT模型的性能。
scGPT模型的验证
文章中,作者为了验证模型scGPT的有效性,设计了多个下游任务,并使用了不同的方法和指标来衡量模型的表现情况。以下是各个下游任务、衡量方法和指标的总结:
-
单细胞RNA测序(cell type annotation):
- 方法: 使用神经网络分类器对scGPT变换器输出的细胞嵌入进行分类,预测细胞类型。
- 指标: 准确性(Accuracy)、精确度(Precision)、召回率(Recall)、宏F1分数(Macro F1)。
- 意义: 这些指标评估了模型在细胞类型分类任务上的性能,包括整体的准确性、对每个类别的精确度和召回率,以及一个综合考虑类别权重的宏F1分数。
- 结果: scGPT在多个数据集上的表现超过了其他方法,包括准确度、精确度、召回率和宏F1分数。
-
遗传扰动响应预测(Perturbation response prediction):
- 方法: 使用Pearsondelta指标来衡量预测和观察到的扰动后基因表达变化之间的相关性。
- 指标: Pearsondelta、Pearsondelta on differentially expressed genes(DE genes)。
- 意义: 这些指标衡量了模型对于未见过的基因扰动响应的预测能力,特别是在预测最显著变化的基因上的表现。
- 结果: scGPT在预测未见基因扰动响应的任务上取得了最高的分数,并且在所有三个数据集上都优于其他方法。
-
多批次scRNA-seq数据整合(Multi-batch scRNA-seq integration):
- 方法: 使用scGPT进行自监督微调,学习统一的细胞表示以恢复掩蔽的基因表达。
- 指标: AvgBIO分数(包括NMIcell、ARIcell和ASWcell)和AvgBATCH分数(包括ASWbatch和GraphConn)。
- 意义: AvgBIO分数衡量了数据整合后细胞类型聚类与真实标签的一致性,而AvgBATCH分数衡量了批次效应校正的性能。
- 结果: scGPT在PBMC 10k数据集上成功分离了所有细胞类型,并且在AvgBIO分数上比比较方法高出5-10%。
-
单细胞多组学(scMultiomic)数据整合:
- 方法: 对于配对和马赛克数据整合设置,scGPT与Seurat (v.4)、scGLUE和scMoMat等方法进行比较。
- 指标: NMIcell、ARIcell、ASWcell、AvgBIO以及ASWbatch、GraphConn、AvgBATCH。
- 意义: 这些指标评估了模型在整合不同组学数据时保持生物学信号和校正批次效应的能力。
- 结果: scGPT在10x Multiome PBMC数据集上成功生成了CD8+ naive细胞的独特聚类,并且在BMMC数据集上展现了更明确的聚类结构。
-
基因调控网络推断(Gene regulatory network inference):
- 方法: 使用scGPT嵌入和注意力图来推断基因调控网络。
- 指标: 未明确提及,但通过与已知生物学和功能组的一致性来验证网络的质量。
- 意义: 这些分析评估了scGPT在推断基因间复杂关系和特定细胞状态下的基因调控网络的能力。
- 结果: scGPT能够通过学习到的基因嵌入和注意力图来揭示生物学上有意义的基因调控网络。
在所有这些任务中,scGPT模型都展现出了优越的性能,这表明了其在单细胞多组学数据建模方面的有效性和潜力。
scGPT验证指标的表现
在文章中,作者通过多个下游任务验证了scGPT模型的有效性,并在各项任务和指标上展示了其出色的表现:
-
单细胞RNA测序数据的细胞类型注释:
- 表现: scGPT在人类胰腺数据集上实现了高精确度(>0.8),并且在多边形混淆矩阵中,大多数细胞类型的预测结果准确。在疾病数据集(如多发性硬化症MS)上,scGPT模型在细胞类型注释上达到了约0.85的高准确率。在肿瘤浸润性髓系数据集上,scGPT在区分免疫细胞亚型方面表现出高精确度。
-
遗传扰动响应预测:
- 表现: 在三个Perturb-seq数据集上评估scGPT的扰动预测能力,scGPT在预测未见基因的扰动响应方面表现优异,通过Pearsondelta指标衡量,scGPT在所有数据集上都取得了最高分,特别是在预测扰动后变化的基因表达上,持续优于其他方法5-20%。
-
多批次scRNA-seq数据整合:
- 表现: 在COVID-19、PBMC 10k和周围皮层数据集上的整合性能评估中,scGPT在细胞类型聚类和批次效应校正方面展现出了优越的整合性能,AvgBIO分数比其他方法高出5-10%。
-
单细胞多组学数据整合:
- 表现: 在10x Multiome PBMC数据集上,scGPT是唯一成功生成CD8+ naive细胞独特聚类的方法。在BMMC数据集上,scGPT比Seurat (v.4)展现出更明确的聚类结构,并且在AvgBIO分数上提高了9%。
-
基因调控网络推断:
- 表现: scGPT能够成功识别与T细胞激活相关的基因群组,如编码T3复合体的CD3基因群,以及与B细胞信号传导和HLA类I分子的共受体相关的基因。在免疫人类数据集上,scGPT模型突出显示了与特定免疫细胞类型相关的CD基因网络。
总体而言,scGPT在所有这些任务中都展现出了卓越的性能,不仅在精确度、准确率、召回率和F1分数等指标上超越了其他方法,而且还能够揭示生物学上有意义的基因调控网络,证明了其在单细胞生物学和遗传研究中的潜力和应用价值。
scGPT模型验证详情
| 任务类型 | 任务描述 | 模型表现 | 评估指标 | 数据集 | 基准/对比方法 |
|---|---|---|---|---|---|
| 细胞类型注释 | 对单细胞RNA测序数据进行细胞类型的分类和注释 | 在人类胰腺数据集上实现高精确度(>0.8);在MS数据集上达到约0.85的准确率 | 准确性(Accuracy)、精确度(Precision)、召回率(Recall)、宏F1分数(Macro F1) | 多发性硬化症(MS)数据集、人类胰腺数据集、骨髓瘤(Myeloid)数据集 | scBERT、TOSICA |
| 遗传扰动响应预测 | 预测基因扰动后的细胞响应 | 在三个Perturb-seq数据集上持续优于其他方法5-20% | Pearsondelta、Pearsondelta on DE genes | Adamson、Norman、Replogle数据集 | GEARS、线性回归模型 |
| 多批次scRNA-seq数据整合 | 整合来自不同批次的单细胞RNA测序数据 | 在PBMC 10k数据集上成功分离所有细胞类型,AvgBIO分数比其他方法高出5-10% | AvgBIO分数(NMIcell、ARIcell、ASWcell)、AvgBATCH分数(ASWbatch、GraphConn) | COVID-19数据集、PBMC 10k数据集、周围皮层数据集 | Seurat、Harmony、scVI |
| 单细胞多组学数据整合 | 整合单细胞多组学数据 | 在10x Multiome PBMC数据集上成功生成CD8+ naive细胞独特聚类;在BMMC数据集上展现更明确聚类结构 | NMIcell、ARIcell、ASWcell、AvgBIO、ASWbatch、GraphConn、AvgBATCH | 10x Multiome PBMC数据集、BMMC数据集、ASAP PBMC数据集 | Seurat (v.4)、scGLUE、scMoMat |
| 基因调控网络推断 | 推断基因间的调控网络 | 能够揭示生物学上有意义的基因调控网络 | 未明确提及,通过与已知生物学和功能组的一致性来验证 | 免疫人类(Immune Human)数据集 | 基于表达相关性的共表达网络 |
scGPT模型验证中使用的数据集和基准/对比方法
| 数据集 | 描述 | 文献引用 |
|---|---|---|
| CELLxGENE scRNA-seq数据集 | 用于构建全人类基础模型的预训练 | cellxgene |
| MS数据集 | 多发性硬化症相关的单细胞RNA测序数据集 | E-HCAD-35 |
| Myeloid数据集 | 骨髓瘤相关的单细胞RNA测序数据集 | GSE154763 |
| Human Pancreas数据集 | 人类胰腺细胞的单细胞RNA测序数据集 | TOSICA |
| PBMC 10k数据集 | 人类PBMC细胞的单细胞RNA测序数据集 | scVI tools |
| Perirhinal Cortex数据集 | 来自人脑皮层的单细胞数据集 | CELLxGENE Human Brain Cell Atlas |
| COVID-19数据集 | 与COVID-19相关的单细胞RNA测序数据集 | scarches-reproducibility |
| Adamson数据集 | CRISPR干扰的基因表达数据集 | 哈佛数据集 |
| Norman数据集 | Perturb-seq技术得到的基因表达数据集 | 哈佛数据集 |
| Replogle数据集 | K562白血病细胞系的基因组范围扰动数据 | MIT数据集 |
| 10x Multiome PBMC数据集 | 单细胞多组学数据集 | scGLUE |
| BMMC数据集 | 包含RNA和蛋白的单细胞多组学数据集 | GSE194122 |
| ASAP PBMC数据集 | 包含多种数据模态的单细胞数据集 | scMoMaT |
| Immune Human数据集 | 用于基因调控网络推断的单细胞数据集 | Figshare |
| 基准/对比方法 | 描述 | 评估指标 | 文献引用 |
|---|---|---|---|
| scBERT | 用于细胞类型注释的基准方法 | 准确性(Accuracy)、精确度(Precision)、召回率(Recall)、宏F1分数(Macro F1) | scBERT |
| TOSICA | 用于细胞类型注释的基准方法 | 同上 | TOSICA |
| GEARS | 遗传扰动响应预测的对比方法 | Pearsondelta 和 Pearsondelta on differentially expressed genes (DE genes) | GEARS |
| Seurat | 多批次scRNA-seq数据整合的对比方法 | AvgBIO分数(包括NMIcell、ARIcell和ASWcell)、AvgBATCH分数(包括ASWbatch和GraphConn) | Seurat |
| Harmony | 多批次scRNA-seq数据整合的对比方法 | 同上 | Harmony |
| scVI | 多批次scRNA-seq数据整合的对比方法 | 同上 | scVI |
| scGLUE | 单细胞多组学数据整合的对比方法 | NMIcell、ARIcell、ASWcell、AvgBIO以及ASWbatch、GraphConn、AvgBATCH | scGLUE |
| scMoMat | 单细胞多组学数据整合的对比方法 | 同上 | scMoMat |
| coexpression network | 基因调控网络推断的基准方法 | 未明确提及,但通过与已知生物学和功能组的一致性来验证 | coexpression network |