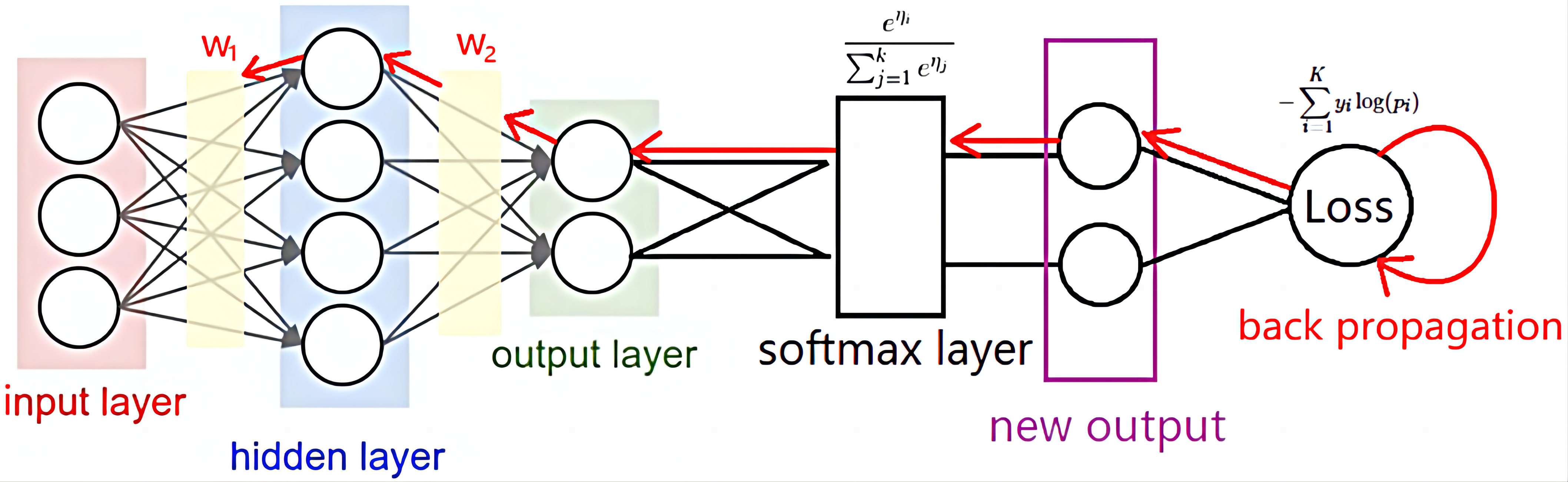
genePT
这篇文章介绍了一个名为GenePT的模型,该模型利用大型语言模型(如GPT-3.5)来生成基因和细胞的嵌入表示,以便于单细胞生物学分析。作者设计了一系列下游任务来验证GenePT模型的有效性,并与现有的单细胞基础模型(如Geneformer和scGPT)进行了比较。以下是文章中提到的下游任务、衡量方法、指标以及数据集的总结:
模型复杂度
scGPT模型的具体参数数量没有直接提及。然而,文章中提到了模型的一些架构细节,例如:
- 预训练基础模型: 嵌入大小为512,包含12个堆叠的变换器块(transformer blocks),每个块有8个注意力头(attention heads)。
- 全连接层: 隐藏层大小为512。
下游任务:
- 基因功能类别预测:预测基因属于的最常见的15个功能类别之一。
- 基因属性预测:包括区分剂量敏感与不敏感的转录因子、区分不同的基因甲基化状态等。
- 基因-基因相互作用预测:预测两个基因是否相互作用。
- 蛋白质-蛋白质相互作用预测:预测两个蛋白质之间是否存在相互作用。
- 细胞类型注释:基于细胞的嵌入表示预测细胞的类型。
- 批次效应评估:评估嵌入表示在去除批次效应的同时保留生物学信息的能力。
衡量方法和指标:
- 准确率(Accuracy):正确预测的数量除以总预测数量。
- 精确度(Precision):正确预测为正的数量除以预测为正的总数量。
- 召回率(Recall):正确预测为正的数量除以实际为正的总数量。
- F1分数:精确度和召回率的调和平均值,衡量模型的准确性和完整性的平衡。
- ROC-AUC:接收者操作特征曲线下的面积,衡量分类器在所有可能的分类阈值上的性能。
- Adjusted Rand Index (ARI) 和 Adjusted Mutual Information (AMI):评估聚类结果与真实标签之间的一致性。
作者的指标表现:
文章中提到GenePT在多个任务上的表现与Geneformer和其他模型相当,甚至在一些任务上表现更好。
Gene功能类别预测:
- GenePT:整体准确率达到96%,类具体准确度和只有小幅度的误分类。
基因-基因相互作用预测 (GGI):
- GenePT:使用共享基因本体 (GO) 注释的测试GGI数据集,ROC-AUC为0.82。
蛋白质-蛋白质相互作用预测 (PPI):
- GenePT:在三个不同的PPI数据集上,ROC-AUC分别为:
- 文献衍生数据集:ROC-AUC未明确给出,但提到表现优于其他模型。
- 综合检测数据集:ROC-AUC未明确给出,但提到表现优于其他模型。
- 组织特异性蛋白质-蛋白质功能网络数据集:ROC-AUC未明确给出,但提到表现优于其他模型。
细胞类型注释:
- GenePT-w 和 GenePT-s 在多个数据集上的表现与 scGPT 和 Geneformer 相比较:
- Aorta 数据集:
- GenePT-w: ARI=0.12, AMI=0.12, ASW=0.01
- GenePT-s: ARI=0.09, AMI=0.12, ASW=-0.04
- Artery 数据集:
- GenePT-w: ARI=0.47, AMI=0.64, ASW=0.18
- GenePT-s: ARI=0.36, AMI=0.59, ASW=0.15
- Bones 数据集:
- GenePT-w: ARI=0.12, AMI=0.21, ASW=-0.01
- GenePT-s: ARI=0.21, AMI=0.29, ASW=0.02
- Myeloid 数据集(癌症类型):
- GenePT-w: ARI=0.25, AMI=0.27, ASW=0.02
- GenePT-s: ARI=0.17, AMI=0.17, ASW=0.06
- Pancreas 数据集:
- GenePT-w: ARI=0.49, AMI=0.69, ASW=0.15
- GenePT-s: ARI=0.30, AMI=0.50, ASW=0.10
- Multiple Sclerosis 数据集(年龄):
- GenePT-w: ARI=0.07, AMI=0.13, ASW=-0.07
- GenePT-s: ARI=0.06, AMI=0.12, ASW=-0.03
- Aorta 数据集:
批次效应评估:
- GenePT 在去除批次效应的同时保留生物学信息的能力表现良好,与 scGPT 和 Geneformer 相比,在多个数据集上的ARI值降低,表明批次效应较小。
ARI和AMI用来衡量聚类结果与真实标签之间的一致性,ASW用来评估聚类结果的凝聚度和分离度。
GenePT在多数任务中展现出与现有模型相当的或更好的性能。
数据集:
- 基因功能类别预测:使用特定的基因功能类别数据集(geneformer)。
- 基因属性预测:使用Theodoris等人提供的开源数据。
- 基因-基因相互作用预测:使用Du等人基于共享基因本体注释的基准数据集。
- 蛋白质-蛋白质相互作用预测:使用了多个数据集,包括HuRI、Lit-BM和Greene等人提供的组织特异性蛋白质-蛋白质功能网络。
- 细胞类型注释:使用了来自循环系统(Aorta、Artery)、骨组织(Bones、Myeloid)、胰腺和来自健康个体以及多发性硬化症患者的免疫细胞的数据集。
基准方法和对比方法:
文章中提到了Geneformer和scGPT作为基准方法。这些方法在不同的任务和指标上的表现被用来与GenePT进行比较。具体的数值表现和比较分析可能在文章的实验部分有详细说明。
总结
| 任务类型 | 指标 | GenePT表现 | 数据集名称 | 基准/对比方法 | 引用链接 |
|---|---|---|---|---|---|
| Gene Functionality Class Prediction | Accuracy | 96% | - | Geneformer等 | - |
| Gene Property Prediction Tasks | Various | 未具体说明 | - | Gene2vec, scGPT | Theodoris et al. 2023 |
| Gene-Gene Interaction Prediction | ROC-AUC | 0.82 | GEO expression data | Gene2vec/scGPT/Geneformer | Du et al. 2019 |
| Protein-Protein Interaction Prediction | ROC-AUC | 未明确给出,但优于其他模型 | HuRI, Lit-BM, Tissue-specific PPI networks | Other models | HuRI dataset, Lit-BM dataset, Tissue-specific PPI networks |
| Cell Type Annotation | ARI, AMI, ASW | 根据不同数据集而异 | Aorta, Artery, Bones等 | scGPT, Geneformer | Chaffin et al. 2022, Li et al. 2020 |
| Batch Effect Assessment | ARI | 降低,表明批次效应较小 | Cardiomyocyte dataset, Aorta dataset | Geneformer, scGPT | Chaffin et al. dataset, Li et al. dataset |
本博客所有文章除特别声明外,均采用 CC BY-NC-SA 4.0 许可协议。转载请注明来自 蒟蒻薯!